
详细介绍:
miRBase Database
microRNA数据库完整介绍与使用攻略
全球最权威的microRNA序列数据库,收录200多个物种的近4万个miRNA信息, 为microRNA研究提供全面的序列、注释和功能信息。
数据库概述
什么是miRBase?
miRBase是由英国曼彻斯特大学维护的全球最权威的microRNA数据库,始建于2002年。 作为microRNA序列和注释的一级数据库,它为全世界的研究人员提供了标准化的miRNA命名、 序列信息和功能注释,是microRNA研究领域不可或缺的重要资源。
数据库规模
发展历程与重要里程碑
miRBase数据库建立
最初命名为MicroRNA Registry,开始收录第一批microRNA序列
重大更新
正式更名为miRBase,建立标准化的miRNA命名规范
版本21发布
引入深度测序数据验证,提高数据可靠性
最新版本22
当前稳定版本,包含最全面的miRNA数据集
核心功能特色
多维度检索
- • 通过miRNA名称/ID检索
- • 基因组位置检索
- • 序列BLAST比对检索
- • 物种特异性浏览
- • 关键词全文检索
序列信息
- • 前体miRNA序列(pre-miRNA)
- • 成熟miRNA序列
- • 茎环二级结构预测
- • 序列保守性分析
- • 同源序列比较
基因组定位
- • 染色体位置信息
- • 基因组坐标
- • 链方向标注
- • 基因组注释
- • 进化保守性
表达数据
- • RPM表达量数据
- • 组织特异性表达
- • 发育阶段表达
- • 疾病相关表达
- • 深度测序验证
靶基因预测
- • 实验验证靶基因
- • 计算预测靶基因
- • 多种预测算法整合
- • 靶基因功能注释
- • 通路富集分析
数据下载
- • FASTA格式序列文件
- • GFF3格式注释文件
- • 表达量数据文件
- • 系统发育树文件
- • 批量数据导出
界面详细介绍
主页面布局解析
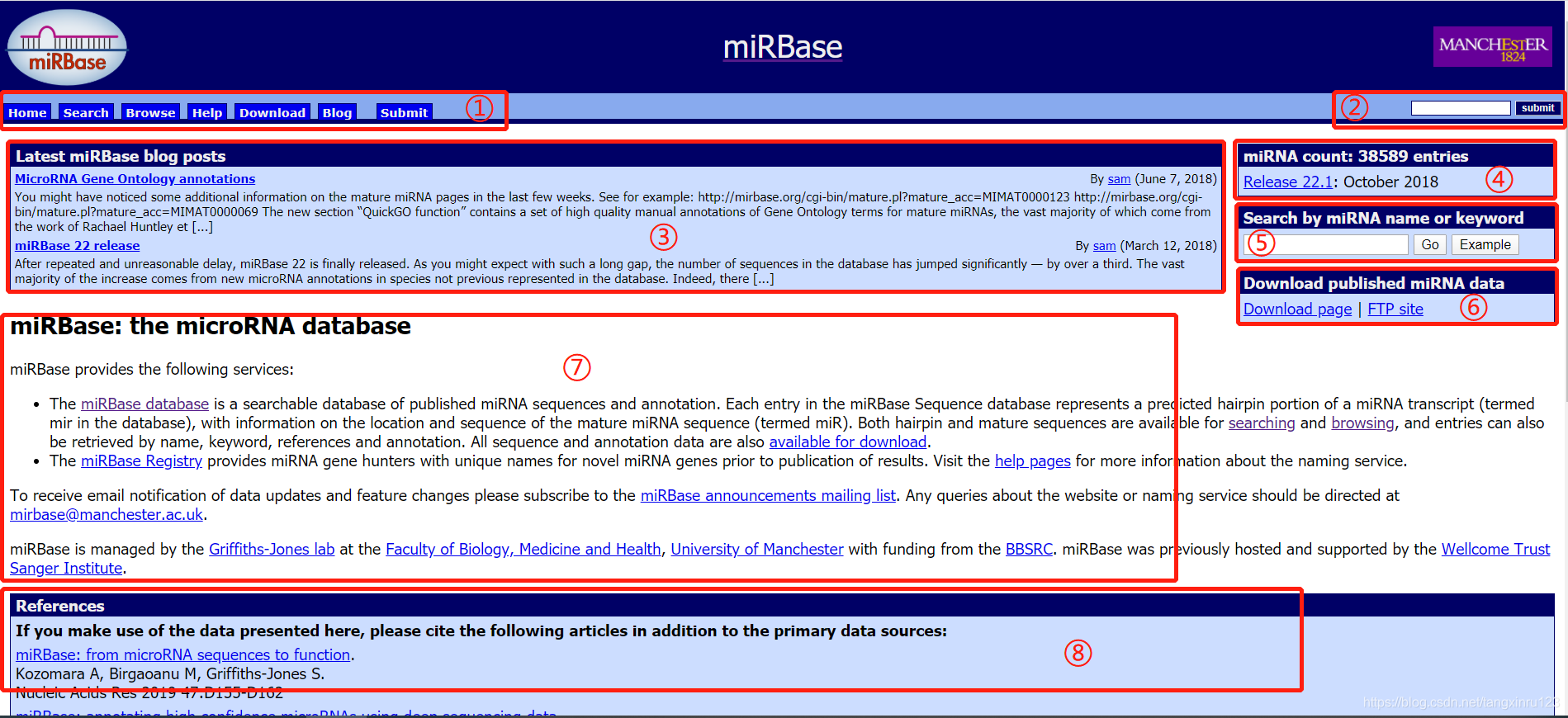
miRBase数据库主页面界面
界面元素说明
包含Home、Search、Browse、Help、Downloads等主要功能模块
支持miRNA名称、ID、关键词等多种检索方式
显示当前版本号、物种数量、miRNA条目数等关键统计信息
检索方式
通过miRNA名称或相关关键词快速查找
基于染色体位置信息进行精确定位
分物种查看所有已知的miRNA信息
功能模块详解
Search(检索)
提供多种检索方式,支持模糊匹配和精确查找
Browse(浏览)
按物种分类浏览,方便系统性查看数据
Downloads(下载)
提供多种格式的数据文件下载
Help(帮助)
详细的使用说明和FAQ解答
页面信息栏
数据库版本信息
显示当前数据库版本号和最后更新时间
统计数据
实时显示数据库中的物种数量、miRNA条目数等
快速链接
提供常用功能和热门数据的快速访问入口
详细检索方法
方法一:通过标识符或关键词检索
支持的检索类型
检索技巧
示例检索词:
- •
hsa-let-7a- 查找人类let-7a家族 - •
let-7*- 使用通配符查找所有let-7 - •
mir-21- 查找miR-21相关序列 - •
MI000*- 查找特定accession范围
方法二:基因组位置检索
输入格式要求
标准格式:
物种:染色体:起始位置:终止位置:链
示例:
- •
hsa:1:1000000:2000000:+ - •
mmu:X:50000000:60000000:- - •
dme:2L:1000000:1100000
支持的物种代码
方法三:按物种浏览
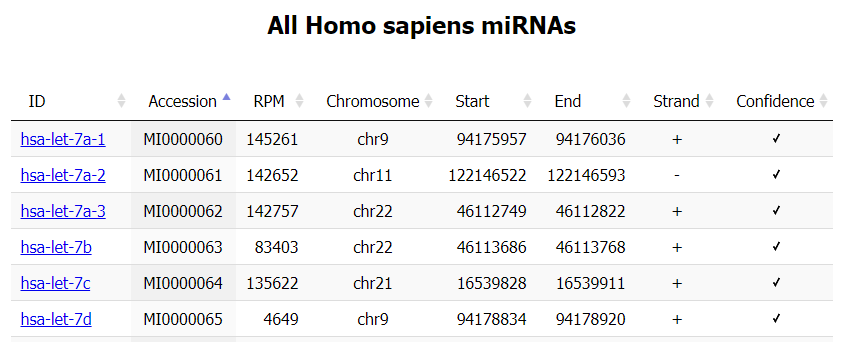
人类miRNA列表浏览界面
列表字段说明
浏览操作技巧
方法四:序列比对检索
BLAST检索
支持的序列格式:
- • FASTA格式序列
- • 原始核酸序列
- • 支持模糊碱基(N, R, Y等)
- • 最大长度:10,000 bp
参数设置
详细使用教程
步骤详解:查找特定miRNA信息
访问miRBase主页
在浏览器中输入 http://www.mirbase.org
小提示:建议使用Chrome或Firefox浏览器以获得最佳体验
选择检索方式
根据你的需求选择以下检索方式之一:
已知miRNA名称
直接在搜索框输入miRNA ID
浏览特定物种
点击Browse按物种查看
输入检索条件
以查找人类let-7a-1为例:
在搜索框中输入:hsa-let-7a-1
或者在Browse页面选择:Homo sapiens
查看检索结果
系统会显示匹配的miRNA列表,点击感兴趣的条目
解读详细信息

miRNA详细信息页面示例
结果页面信息解读
基本信息区域
miRNA ID
标准化的miRNA命名标识符
Accession
数据库唯一登录号
Description
miRNA的描述性信息
序列信息区域
Sequence
前体miRNA的完整序列
Structure
茎环二级结构预测图
Mature sequences
成熟miRNA序列(5p和3p)

成熟miRNA序列和靶基因信息
数据下载指南
FTP下载站点
ftp://mirbase.org/pub/mirbase/CURRENT/
miRBase官方FTP下载地址,包含最新版本的所有数据文件
hairpin.fa
所有miRNA前体序列的FASTA格式文件
mature.fa
所有成熟miRNA序列的FASTA格式文件
miRNA.dat
详细的miRNA注释信息数据文件
genomes/
按物种分类的基因组坐标文件
miRNA_RPM.tsv
miRNA表达量数据(RPM值)
families/
miRNA家族分类和系统发育信息
命令行下载示例
下载所有前体序列:
wget ftp://mirbase.org/pub/mirbase/CURRENT/hairpin.fa.gz
下载人类miRNA数据:
wget ftp://mirbase.org/pub/mirbase/CURRENT/genomes/hsa.gff3
批量下载:
wget -r ftp://mirbase.org/pub/mirbase/CURRENT/数据筛选技巧
按物种筛选
使用grep命令筛选特定物种的miRNA:
grep "^>hsa" hairpin.fa提取序列
使用Python或R脚本提取特定miRNA序列:
seqkit grep -p "let-7" hairpin.fa格式转换
转换为不同的序列格式:
seqkit fq2fa input.fastq > output.fasta实用技巧与建议
高效检索技巧
- • 使用通配符(*)进行模糊匹配
- • 善用物种代码缩写检索
- • 结合关键词和ID精确查找
- • 利用浏览器书签保存常用检索
- • 记录重要miRNA的Accession号
数据管理建议
- • 定期更新本地miRNA数据库
- • 建立个人miRNA研究档案
- • 使用版本控制管理序列文件
- • 备份重要的检索结果
- • 记录数据来源和版本信息
分析工具推荐
- • TargetScan:靶基因预测
- • miRTarBase:实验验证靶基因
- • RNAfold:二级结构预测
- • miRDeep2:新miRNA发现
- • DIANA-Tools:综合分析套件
常见问题解决
- • 网络超时:使用FTP镜像站点
- • 序列不匹配:检查物种代码
- • 版本差异:确认数据库版本
- • 命名问题:参考官方命名规范
- • 下载失败:分批下载小文件
学习资源
- • miRBase官方帮助文档
- • Nature Methods相关论文
- • Bioinformatics在线课程
- • GitHub开源分析脚本
- • 学术会议workshop资料
社区支持
- • miRBase官方邮件列表
- • Biostars问答社区
- • ResearchGate讨论组
- • Stack Overflow标签
- • 学术Twitter关注
最佳实践建议
研究流程优化
前期准备
明确研究物种和miRNA家族,制定检索策略
数据收集
系统性收集相关miRNA信息,建立本地数据库
质量控制
验证序列信息准确性,交叉比对多个数据源
数据引用规范
标准引用格式
按照期刊要求正确引用miRBase数据库
版本标注
明确标注使用的miRBase版本号和访问日期
数据可重现性
提供详细的数据处理方法和参数设置
总结与展望
权威性
miRBase作为全球标准的miRNA数据库,为科研提供可靠的数据支撑
全面性
覆盖200多个物种的近4万个miRNA,满足各种研究需求
持续性
定期更新维护,紧跟miRNA研究的最新进展
miRBase数据库是miRNA研究领域不可或缺的重要资源。掌握其使用方法, 能够显著提高研究效率,为深入理解miRNA的功能和调控机制提供坚实基础。
参考文献与相关资源
核心文献
Kozomara et al. (2019)
miRBase: from microRNA sequences to function. Nucleic Acids Res.
Griffiths-Jones et al. (2006)
miRBase: microRNA sequences, targets and gene nomenclature. Nucleic Acids Res.
Kozomara & Griffiths-Jones (2014)
miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Res.

 执业医师资格考试培训
执业医师资格考试培训 免费试听
免费试听 执业药师考试培训
执业药师考试培训



